2025年2月,我院生物进化团队张代臻教授团队在国际知名期刊 BMC Biology上发表了题为“The chromosome-level genome provides insights into the adaptive evolution of the visual system in Oratosquilla oratoria”的研究论文。该研究填补了国际上口足目类群物种基因组的空白,并且为理解甲壳类动物视觉系统的多样性和复杂性提供了新的视角,有利于口虾蛄的保护和可持续利用。盐城师范学院湿地学院生物进化研究团队张代臻教授、已毕业生孙晓丽(广西大学博士)、华中农业大学陈连福、福建农林大学林联宇为共同一作,湿地学院王刚副教授、唐伯平教授、张代臻教授和西北工业大学李永鑫教授为本文的共同通讯作者。该研究得到国家自然基金项目支持。

口虾蛄(Oratosquilla oratoria),又名螳螂虾,是一种具有重要经济价值的海洋甲壳类动物,广泛分布于中国、日本和韩国沿海水域。虾蛄以鲜艳的色彩、独特的身体结构和卓越的捕食能力而闻名,其高度发达的视觉系统使其能够感知广泛的色觉和偏振光。然而,由于缺乏高质量的基因组组装,人们对口虾蛄视觉系统发育的分子机制知之甚少。

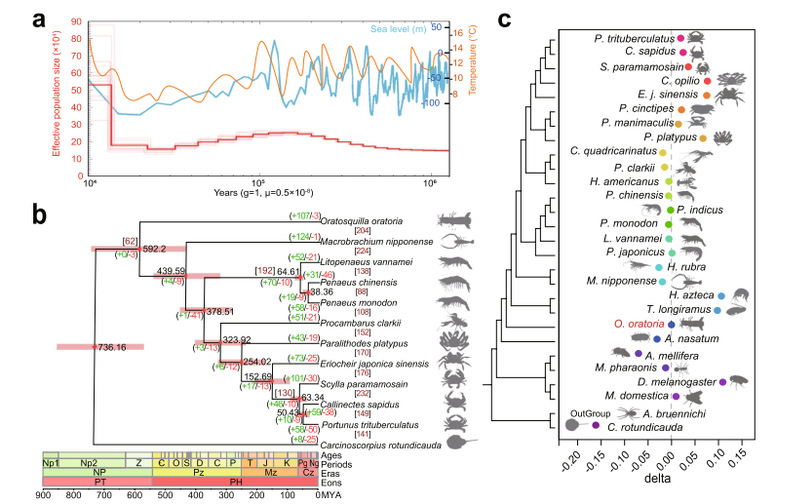
该研究利用 PacBio 长读测序、Illumina 短读测序和 Hi-C 技术,成功构建了口虾蛄的染色体水平基因组,基因组大小为 2.97 Gb,包含 44 条伪染色体。基因组的 Contig N50 达到 1.66 Mb,Scaffold N50 为 65.98 Mb,整体锚定率约为 95.27%。BUSCO 评估显示基因组完整性为 90.3%,预测出 30831 个基因模型。口虾蛄基因组中超过一半的区域被重复序列覆盖,其中长散在核元件(LINE)占比高达 19.58%,显著高于其他类型的重复序列。口虾蛄基因组中近期(约 0.17-0.27 百万年前)发生了大规模的长末端重复序列(LTR)扩张,这一现象在其他海洋甲壳类动物中也有所观察,表明这些物种可能在该时期经历了剧烈的环境变化。

通过系统发育分析,研究者发现口虾蛄与十足目(包括对虾亚目和真虾亚目)关系密切,而与剑尾目(Merostomata)的马蹄蟹(Carcinoscorpius rotundicauda)作为外类群时,口虾蛄与十足目明显分离。基于估计的分歧时间,口虾蛄与小龙虾(Cambarus)和蟹类的共同祖先大约在 5.922 亿年前分化,而对虾亚目和真虾亚目大约在 3.785 亿年前分化。此外,研究还发现口虾蛄的进化速率介于对虾亚目和真虾亚目之间,这可能与它们不同的生态位和环境条件有关。
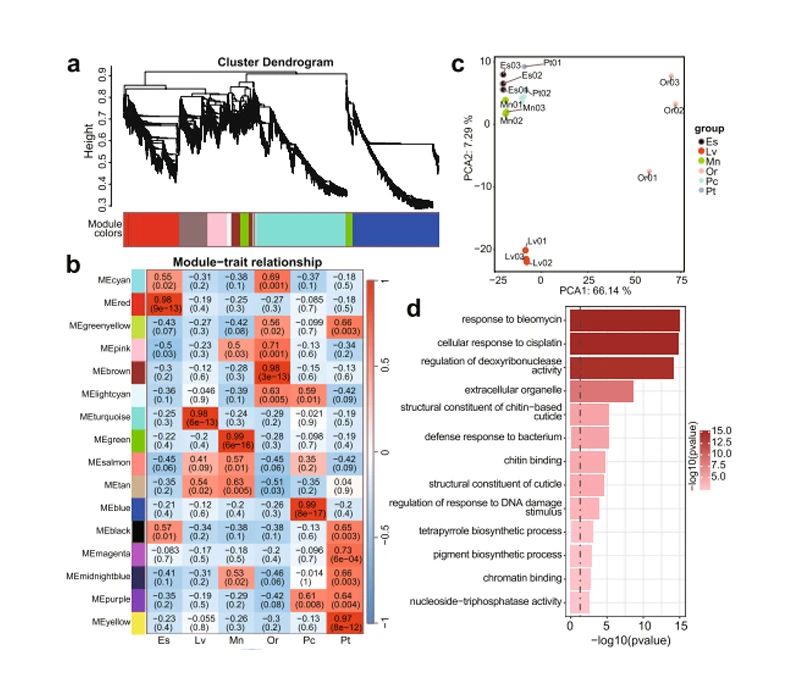
视蛋白基因家族扩张:口虾蛄基因组中鉴定出 34 个视蛋白基因,包括 7 个长波长敏感(LWS)视蛋白基因、26 个中波长敏感(MWS)视蛋白基因和 1 个短波长敏感(SWS)视蛋白基因。其中,MWS 视蛋白基因通过串联重复显著扩张,形成了三个独立的基因簇,分别命名为 MWS-I、MWS-II 和 MWS-III,其中 MWS-I 和 MWS-II 显著扩张,而 MWS-III 在口虾蛄基因组中丢失。为了验证 MWS 视蛋白基因的功能,研究者将口虾蛄暴露于绿光(520-530 nm 波长)下 12 小时,发现 5 个 MWS 视蛋白基因显著激活,而在可见光谱下几乎不表达,这表明口虾蛄的 MWS 视蛋白可能在特定生态需求下被激活,如捕食、躲避天敌或选择配偶。这一结果也表明,口虾蛄的视觉系统可能经过特殊进化,以适应其在浅海环境中对绿光的高敏感性。

角膜相关基因鉴定:口虾蛄具有比其他甲壳类动物更厚的角膜,研究者通过比较转录组分析,发现一个串联重复的几丁质蛋白(CP)基因簇在口虾蛄眼组织中特异性扩张并表达。该 CP 基因簇包含 16 个 CP 基因,分布在约 203 kb 的基因组区域中。通过系统发育分析,研究者发现该 CP 基因簇在口虾蛄基因组中特异性扩张,且仅在口虾蛄的眼组织中表达,而在其他甲壳类动物中未检测到表达。角膜的增厚有助于优化光的折射,提高视觉清晰度,同时为口虾蛄提供更强的物理保护,使其能够在捕食和防御时减少角膜损伤的风险。